| Labfans是一个针对大学生、工程师和科研工作者的技术社区。 | 论坛首页 | 联系我们(Contact Us) |
 |
| Labfans是一个针对大学生、工程师和科研工作者的技术社区。 | 论坛首页 | 联系我们(Contact Us) |
 |
|
|
#1 |
|
高级会员
注册日期: 2019-11-21
帖子: 3,020
声望力: 67  |
在Matlab中,我想绘制一个氨基酸序列图的seqlogo图 。但是,我不希望通过熵来缩放绘图列的高度,而是希望所有列都具有相同的高度。
我正在根据该问题的答案来修改代码,但我想知道seqlogo的参数或其他我遗漏的其他函数是否可以使列高保持一致。 另外,是否可以对序列配置文件进行统计转换以破解所需的输出? (列高均匀,每个字母的高度与其在seqprofile中的概率成线性比例) 回答: 解决此问题的最简单方法可能是直接修改生物信息学工具箱功能SEQLOGO的代码(如果可能)。在R2010b中,您可以执行以下操作: edit seqlogo 该功能的代码将显示在编辑器中。接下来,找到以下几行(第267-284行)并将其注释掉或将其完全删除: S_before = log2(nSymbols); freqM(freqM == 0) = 1; % log2(1) = 0 % The uncertainty after the input at each position S_after = -sum(log2(freqM).*freqM, 1); if corrError % The number of sequences correction factor e_corr = (nSymbols -1)/(2* log(2) * numSeq); R = S_before - (S_after + e_corr); else R = S_before - S_after; end nPos = (endPos - startPos) + 1; for i =1:nPos wtM(:, i) = wtM(:, i) * R(i); end 然后将此行放在他们的位置: wtM = bsxfun(@times,wtM,log2(nSymbols)./sum(wtM)); 您可能希望将文件保存为新名称,例如seqlogo_norm.m ,因此您仍然可以使用原始的未修改SEQLOGO函数。现在,您可以创建序列轮廓图,将所有列标准化为相同的高度。例如: S = {'LSGGQRQRVAIARALAL',... %# Sample amino acid sequence 'LSGGEKQRVAIARALMN',... 'LSGGQIQRVLLARALAA',... 'LSGGERRRLEIACVLAL',... 'FSGGEKKKNELWQMLAL',... 'LSGGERRRLEIACVLAL'}; seqlogo_norm(S,'alphabet','aa'); %# Use the modified SEQLOGO function 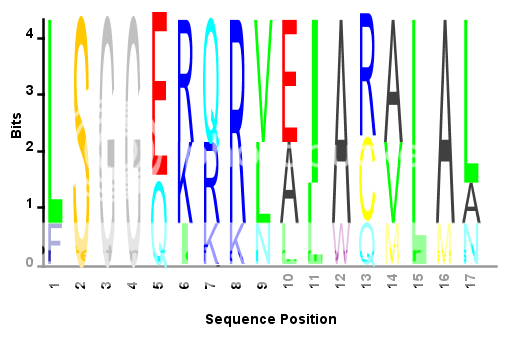 旧答案: 我不确定如何转换序列配置文件信息以从Bioinformatics Toolbox函数SEQLOGO中获得所需的输出,但是我可以向您展示如何修改为我对链接到的相关问题的回答而编写的替代seqlogo_new.m 。如果您bitValues更改用于初始化bitValues的行: bitValues = W{2}; 对此: bitValues = bsxfun(@rdivide,W{2},sum(W{2})); 然后,您应该将每列缩放为1的高度。例如: S = {'ATTATAGCAAACTA',... %# Sample sequence 'AACATGCCAAAGTA',... 'ATCATGCAAAAGGA'}; seqlogo_new(S); %# After applying the above modification  更多&回答... |
|
|

|